- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- STATE_tutorial/H2_jp へ行く。
- 1 (2020-10-30 (金) 22:31:25)
- 2 (2020-10-30 (金) 22:45:08)
水素分子 †
このチュートリアルでは分子系についてどのように平面波カットオフとユニットセルの大きさについての収束性を調べるかを説明します。 周期境界条件の下で孤立分子のシミュレーションを行う場合、分子は周期イメージとの相互作用が無視できる程度に大きな箱(ユニットセル)を用います。 ここでは水素分子を取り上げ、局所密度近似を用いて計算を行います。 使用するポテンシャルはpot.H_lda1です。
SCF 計算 †
- 入力ファイル (nfinp_1)
0 0 0 0 0 0 : H2 molecule in a box 5.0 10.0 1 2 2 : GMAX GMAXP NTYP NATM NATM2 1 0 : NUM_SPACE_GROUP TYPE_OF_BRAVIS_LATTICE 10.0 10.0 10.0 90.0 90.0 90.0 : A B C ALPHA BETA GAMMA 1 1 1 1 1 1 : N1 N2 N3 M1 M2 M3 (k-point mesh and shift) 1 0 : NCORD NINV -0.710 0.000 0.000 1 1 1 : CPS(1:3) IWEI IMDTYP ITYP 0.710 0.000 0.000 1 1 1 : CPS(1:3) IWEI IMDTYP ITYP 1 0.15 1.00794 3 1 0.d0 : IATOMN ALFA AMION ILOC IVAN 0 0 0 0 0 : ICOND INIPOS INIVEL ININOS INIACC 0 1 : IPRE IPRI 200 200 0 57200.00 0 : NMD1 NMD2 ITER_LAST CPUMAX IFSTOP 3 1 : MIX_WAY MIX_WHAT 0 8 0.7 : STARTING_MIXING KBXMIX ALPHA 0.60 0.50 0.60 0.70 1.00 : DTIM1 DTIM2 DTIM3 DTIM4 DTIM_LAST 100.0 4 1 0.10D-08 : DTIO IMDALG IEXPL EDELTA 0.0010 0.50D+03 0 : WIDTH FORCCR ISTRESS ldapw91 1 : XCTYPE NSPIN 1.00 : DESTM 101 : NBZTYP 0 0 0 : DUMMY 0 0 0 : DUMMY 2 : NEG 1 : NEXTST 0 : DUMMY 2 : IMSD 0 : EVALUATE_EKO_DIFF 0 : NPDOSAO 0 0.0 : SM_N dopping - 実行
$ mpirun -np 4 ./STATE < nfinp_1 > nfout_1
- 結果
STATEの実行されると、先ず最初に以下のメッセージが表示されます。*********************************************************************** * * * * * * * ****** ******** ** ******** ******** * * ******** ******** **** ******** ******** * * ** ** ** ** ** ** * * *** ** ******** ** ****** * * *** ** ********** ** ****** * * ** ** ** ** ** ** * * ******** ** ** ** ** ******** * * ****** ** VERSION 5.6.5 ** ******** * * RICS-AIST * * OSAKA UNIVERSITY * * * ***********************************************************************
SCF計算が収束すると以下の全エネルギーおよび運動エネルギーなどの寄与が表示されます。TOTAL ENERGY AND ITS COMPONENTS TOTAL ENERGY = -1.13758782 A.U. FREE ENERGY = -1.13758782 A.U. KINETIC ENERGY = 0.96824571 A.U. HARTREE ENERGY = 0.73570075 A.U. XC ENERGY = -0.64555732 A.U. LOCAL ENERGY = -2.31372346 A.U. NONLOCAL ENERGY = -0.02336940 A.U. EWALD ENERGY = 0.14111590 A.U. PC ENERGY = 0.00000000 A.U. ENTROPIC ENERGY = 0.00000000 A.U.全エネルギー以降にフェルミエネルギー、Kohn-Sham固有値と占有数が表示されます。FERMI ENERGY = -0.19018506 NKP= 1 NGP= 2103 K=( 0.00000 0.00000 0.00000) WKP= 1.0000 EIGEN VALUE -0.37074 -0.00963 OCCUPATION 1.00000 0.00000さらに原子にかかる力が以下のように表示されます。ATOM COORDINATES FORCES MD: 1 MD: 1 H -0.710000 0.000000 0.000000 -0.01200 -0.00000 0.00000 MD: 2 H 0.710000 0.000000 0.000000 0.01200 -0.00000 0.00000
収束性の調査 †
平面波基底を用いた計算の場合、波動関数の展開のための平面波カットオフエネルギーが計算の精度を決定する最も重要なパラメーターの一つとなります。 ノルム保存擬ポテンシャルを用い、なおかつ非線形内殻補正を考慮しない場合、電荷密度の平面波カットオフエネルギーは波動関数のそれの4倍になります。 しかしながらウルトラソフト擬ポテンシャルの場合は局在した補強電荷を正確に記述するためにしばしば波動関数についての4倍以上のカットオフエネルギーが必要となる場合があります。 STATEでは波動関数を展開する平面波のカットオフ波数ベクトル (GMAX) を設定しています。
In the STATE code, cutoff wave vectors in the atomic unit for the wave functions (GMAX) and the augmentation charge (GMAXP) should be specified. GMAX**2 and GMAXP**2 are the cutoff energies in Ry.
By performing the total energy calculation with varying GMAX and GMAXP, we obtain the following [GMAXP=15 (E_cut^den=225 Ry)]
#GMAX ETOT (Ha) 4.0 -1.12781490 4.5 -1.13418820 5.0 -1.13697688 5.5 -1.13816998 6.0 -1.13847300 6.5 -1.13850127 7.0 -1.13851867 7.5 -1.13857102
[GMAXP=20 (E_cut^den=400 Ry)]
#GMAX ETOT (Ha) 4.0 -1.12780183 4.5 -1.13417130 5.0 -1.13695809 5.5 -1.13815036 6.0 -1.13845313 6.5 -1.13848136 7.0 -1.13849876 7.5 -1.13855109
[GMAXP=25 (E_cut^den=625 Ry)]
#GMAX ETOT (Ha) 4.0 -1.12780130 4.5 -1.13417074 5.0 -1.13695751 5.5 -1.13814979 6.0 -1.13845255 6.5 -1.13848078 7.0 -1.13849818 7.5 -1.13855052
[GMAXP=30 (E_cut^den=900 Ry)]
#GMAX ETOT (Ha) 4.0 -1.12780182 4.5 -1.13417124 5.0 -1.13695801 5.5 -1.13815027 6.0 -1.13845303 6.5 -1.13848127 7.0 -1.13849867 7.5 -1.13855100
With GMAX=6 and GMAXP=20, the total energy converges better than 0.1 mRy.
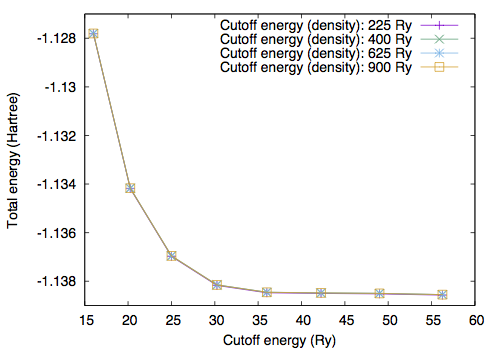
In addition to the cutoff energy, the convergence with respect to the unit cell size should be achieved to simulate an isolated molecule. Using GMAX=6 and GMAXP=20, total energy as a function of cell edge is obtained as follows
#A(Bohr) ETOT(Ha) 5.00 -1.31378028 10.00 -1.13845313 15.00 -1.13662806 20.00 -1.13661187
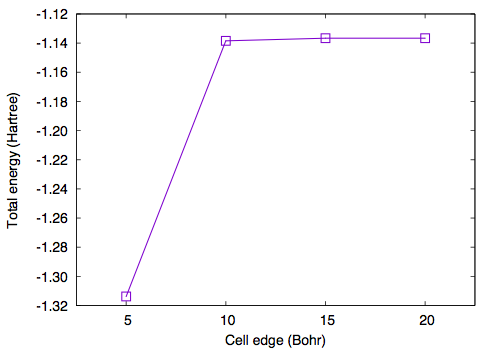
We can see that the cell edge of 10 Bohr is sufficient in the case of an H2 molecule, but a larger box may be needed when larger molecules are studied.
Geometry optimization (1) †
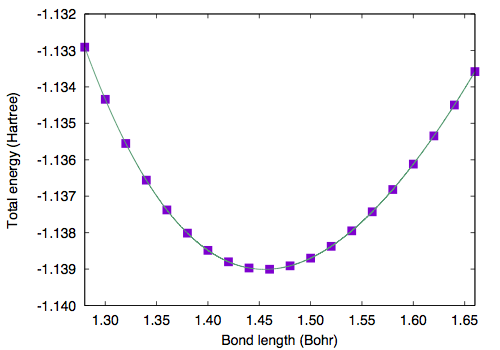
Geometry of a diatomic molecule can be optimized by calculating the total energy with varying bond length. Here the total energy as a function of bond length is shown. GMAX=6, GMAXP=20, and the unit cell edges of 10.0 Bohr is used.
# Bond length (Bohr) Total energy (Hartree) 1.28000000 -1.13291015 1.30000000 -1.13434083 1.32000000 -1.13555229 1.34000000 -1.13655937 1.36000000 -1.13737569 1.38000000 -1.13801370 1.40000000 -1.13848494 1.42000000 -1.13880026 1.44000000 -1.13896985 1.46000000 -1.13900337 1.48000000 -1.13890994 1.50000000 -1.13869810 1.52000000 -1.13837576 1.54000000 -1.13795024 1.56000000 -1.13742831 1.58000000 -1.13681622 1.60000000 -1.13611994 1.62000000 -1.13534513 1.64000000 -1.13449712 1.66000000 -1.13358088
By fitting the total energy to sixth order polynomial, equilibrium bond length of 1.4551 Bohr (0.77 Angstrom).
Geometry optimization (2) †
In a complex system, manual optimization is difficult to perform. In such a case, Hellmann-Feynman forces are used to perform the geometry optimization. To do so, we set the force criterion (FORCCR) of 1.e-3 to 1.e-4 Hartree/Bohr. Note that for SCF calculation, we set a large FORCCR such as 1.e+3 and avoid the structural optimization. In this example, generalized direct inversion of iterative subspace (GDIIS) method is used (IMDALG=4) with the time step of 100 atomic unit.
0 0 0 0 0 0 : H2 molecule in a box
6.0 20.0 1 2 2 : GMAX GMAXP NTYP NATM NATM2
1 0 : NUM_SPACE_GROUP TYPE_OF_BRAVIS_LATTICE
10.0 10.0 10.0 90.0 90.0 90.0 : A B C ALPHA BETA GAMMA
1 1 1 1 1 1 : N1 N2 N3 M1 M2 M3 (k-point mesh and shift)
1 0 : NCORD NINV
-0.710 0.000 0.000 1 1 1 : CPS(1:3) IWEI IMDTYP ITYP
0.710 0.000 0.000 1 1 1 : CPS(1:3) IWEI IMDTYP ITYP
1 0.15 1.00794 3 1 0.d0 : IATOMN ALFA AMION ILOC IVAN
0 0 0 0 0 : ICOND INIPOS INIVEL ININOS INIACC
0 1 : IPRE IPRI
200 200 0 57200.00 0 : NMD1 NMD2 ITER_LAST CPUMAX IFSTOP
3 1 : MIX_WAY MIX_WHAT
0 8 0.7 : STARTING_MIXING KBXMIX ALPHA
0.60 0.50 0.60 0.70 1.00 : DTIM1 DTIM2 DTIM3 DTIM4 DTIM_LAST
100.0 4 1 0.10D-08 : DTIO IMDALG IEXPL EDELTA
0.0010 0.50D-03 0 : WIDTH FORCCR ISTRESS
ldapw91 1 : XCTYPE NSPIN
1.00 : DESTM
101 : NBZTYP
0 0 0 : DUMMY
0 0 0 : DUMMY
2 : NEG
1 : NEXTST
0 : DUMMY
2 : IMSD
0 : EVALUATE_EKO_DIFF
0 : NPDOSAO
0 0.0 : SM_N dopping
Calculated equilibrium bond length is 1.455 Bohr (0.77 Angstrom), agreeing well with the one obtained by the total energy fitting.
Excercise †
- Perform the convergence study of bond length with respect to box size and cutoff energy.
![[PukiWiki] [PukiWiki]](image/logo_state-senri3_80x80.png)