- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- STATE_new_tutorial/Pt111-MetOH へ行く。
- 1 (2021-02-16 (火) 14:27:30)
- 2 (2021-02-16 (火) 17:15:23)
- 3 (2021-02-16 (火) 19:05:51)
メタノール/Pt(111)界面 †
- 目次
- はじめに
このチュートリアルではPt(111)(3x3)表面に吸着したメタノールを例に取りCrystal orbital overlap population (COOP) 解析の方法を説明します。 COOP解析では以下の手順で計算を行います。- 吸着系の構造最適化
- 吸着系のSCF計算
- サブシステム(分子、基板)のSCF計算
- COOP計算の準備(オーバーラップの計算)
- COOP解析
という手順で計算を行います。 また吸着系の構造は既に最適化しているとものとし、吸着系、分子、基板は以下のディレクトリで計算を行うことにします。
CH3OH/ Pt111/ Total/
吸着系のSCF計算 †
- 入力ファイル (Total/nfinp_scf)
#
# CH3OH on Pt(111)
#
RESTART
WF_OPT DAV
NTYP 4
NATM 51
GMAX 6.0
GMAXP 20.0
KPOINT_MESH 4 4 1
WAY_MIX 6
MIX_ALPHA 0.2
SMEARING MP
WIDTH 0.02
EDELTA 1.D-12
NEG 374
&CELL
15.935782493970 0.000000000000 0.000000000000
-7.967891246985 13.800792468961 0.000000000000
0.000000000000 0.000000000000 65.057559603672
&END
&ATOMIC_SPECIES
Pt 195.084 pot.Pt_pbe1
H 1.00784 pot.H_lda1
C 12.0107 pot.C_pbe1
O 15.9994 pot.O_pbe1
&END
&ATOMIC_COORDINATES CARTESIAN
-1.8137028864 9.7577897649 6.0475265334 1 1 3
0.3796625950 9.4143384000 4.4413806754 1 1 4
-2.8866486133 7.9909878630 5.9662288609 1 1 2
1.3575317442 10.9863245180 4.4084228902 1 1 2
-3.0023990778 11.3232130902 5.3771267684 1 1 2
-1.1679860539 10.1062442042 7.9898686733 1 1 2
-0.0099652505 -0.0118152838 -0.0389267708 1 1 1
5.2888683372 0.0108921856 -0.0645303625 1 1 1
10.6265696551 0.0008317009 -0.0387744777 1 1 1
-2.6897091584 4.5448841905 -0.0523847489 1 1 1
2.6635463467 4.5580537376 -0.0706515335 1 1 1
7.9589507735 4.5854784695 -0.0722719517 1 1 1
-5.3637785701 9.1883685551 -0.0662604821 1 1 1
-0.0392907170 9.1565874594 0.0579966667 1 1 1
5.3281043692 9.1911533151 -0.0722007294 1 1 1
-0.0021214349 3.0614887173 -4.4301258499 1 1 1
5.3127589060 3.0682635440 -4.4675412941 1 1 1
10.6216354438 3.0680025298 -4.4594236536 1 1 1
-2.6410480936 7.6736054392 -4.3637257870 1 1 1
2.6463501164 7.6713174600 -4.3821208007 1 1 1
7.9673552854 7.6640546571 -4.4695568010 1 1 1
-5.3175076764 12.2702284809 -4.4320528195 1 1 1
0.0018447605 12.2611966334 -4.3749773596 1 1 1
5.3128659353 12.2716266162 -4.4267739348 1 1 1
0.0011760521 -3.0598724283 -8.7324837135 1 1 1
5.3220998470 -3.0659574817 -8.6963658587 1 1 1
10.6177996176 -3.0665582052 -8.6965151025 1 1 1
-2.6520476401 1.5342773535 -8.7269069007 1 1 1
2.6533798972 1.5333891366 -8.7316179193 1 1 1
7.9713478584 1.5258548152 -8.7275686163 1 1 1
-5.2901369599 6.1501470806 -8.7275606477 1 1 1
0.0020257219 6.1445633683 -8.6976678046 1 1 1
5.3019326567 6.1448522618 -8.7326135025 1 1 1
0.0000000000 0.0000000000 -13.0115119207 1 0 1
5.3119274980 0.0000000000 -13.0115119207 1 0 1
10.6238549960 0.0000000000 -13.0115119207 1 0 1
-2.6559637490 4.6002641563 -13.0115119207 1 0 1
2.6559637490 4.6002641563 -13.0115119207 1 0 1
7.9678912470 4.6002641563 -13.0115119207 1 0 1
-5.3119274980 9.2005283126 -13.0115119207 1 0 1
-0.0000000000 9.2005283126 -13.0115119207 1 0 1
5.3119274980 9.2005283126 -13.0115119207 1 0 1
-0.0000000000 3.0668427709 -17.3486825610 1 0 1
5.3119274980 3.0668427709 -17.3486825610 1 0 1
10.6238549960 3.0668427709 -17.3486825610 1 0 1
-2.6559637490 7.6671069272 -17.3486825610 1 0 1
2.6559637490 7.6671069272 -17.3486825610 1 0 1
7.9678912470 7.6671069272 -17.3486825610 1 0 1
-5.3119274980 12.2673710835 -17.3486825610 1 0 1
-0.0000000000 12.2673710835 -17.3486825610 1 0 1
5.3119274980 12.2673710835 -17.3486825610 1 0 1
&END
&ESM
BOUNDARY_CONDITION BARE
&END
``qsub``を使用、あるいは``mpirun``を用いてSTATEの実行を行います。
吸着分子のSCF計算 †
- 入力ファイル (CH3OH/nfinp_scf)
# # CH3OH # WF_OPT DAV NTYP 4 NATM 6 GMAX 6.0 GMAXP 20.0 KPOINT_MESH 4 4 1 WAY_MIX 6 MIX_ALPHA 0.5 SMEARING MP WIDTH 0.02 EDELTA 1.D-12 NEG 16 &CELL 15.935782493970 0.000000000000 0.000000000000 -7.967891246985 13.800792468961 0.000000000000 0.000000000000 0.000000000000 65.057559603672 &END &ATOMIC_SPECIES Pt 195.084 pot.Pt_pbe1 H 1.00784 pot.H_lda1 C 12.0107 pot.C_pbe1 O 15.9994 pot.O_pbe1 &END &ATOMIC_COORDINATES CARTESIAN -1.8137028864 9.7577897649 6.0475265334 1 1 3 0.3796625950 9.4143384000 4.4413806754 1 1 4 -2.8866486133 7.9909878630 5.9662288609 1 1 2 1.3575317442 10.9863245180 4.4084228902 1 1 2 -3.0023990778 11.3232130902 5.3771267684 1 1 2 -1.1679860539 10.1062442042 7.9898686733 1 1 2 &END &ESM BOUNDARY_CONDITION BARE &END吸着系と同様にSCF計算を実行します。
基板のSCF計算 †
- Pt111/nfinp_scf
# # Pt(111) # WF_OPT DAV NTYP 4 NATM 45 GMAX 6.0 GMAXP 20.0 KPOINT_MESH 4 4 1 NSCF 400 WAY_MIX 6 MIX_ALPHA 0.2 SMEARING MP WIDTH 0.02 EDELTA 1.D-12 NEG 358 &CELL 15.935782493970 0.000000000000 0.000000000000 -7.967891246985 13.800792468961 0.000000000000 0.000000000000 0.000000000000 65.057559603672 &END &ATOMIC_SPECIES Pt 195.084 pot.Pt_pbe1 H 1.00784 pot.H_lda1 C 12.0107 pot.C_pbe1 O 15.9994 pot.O_pbe1 &END &ATOMIC_COORDINATES CARTESIAN -0.0099652505 -0.0118152838 -0.0389267708 1 1 1 5.2888683372 0.0108921856 -0.0645303625 1 1 1 10.6265696551 0.0008317009 -0.0387744777 1 1 1 -2.6897091584 4.5448841905 -0.0523847489 1 1 1 2.6635463467 4.5580537376 -0.0706515335 1 1 1 7.9589507735 4.5854784695 -0.0722719517 1 1 1 -5.3637785701 9.1883685551 -0.0662604821 1 1 1 -0.0392907170 9.1565874594 0.0579966667 1 1 1 5.3281043692 9.1911533151 -0.0722007294 1 1 1 -0.0021214349 3.0614887173 -4.4301258499 1 1 1 5.3127589060 3.0682635440 -4.4675412941 1 1 1 10.6216354438 3.0680025298 -4.4594236536 1 1 1 -2.6410480936 7.6736054392 -4.3637257870 1 1 1 2.6463501164 7.6713174600 -4.3821208007 1 1 1 7.9673552854 7.6640546571 -4.4695568010 1 1 1 -5.3175076764 12.2702284809 -4.4320528195 1 1 1 0.0018447605 12.2611966334 -4.3749773596 1 1 1 5.3128659353 12.2716266162 -4.4267739348 1 1 1 0.0011760521 -3.0598724283 -8.7324837135 1 1 1 5.3220998470 -3.0659574817 -8.6963658587 1 1 1 10.6177996176 -3.0665582052 -8.6965151025 1 1 1 -2.6520476401 1.5342773535 -8.7269069007 1 1 1 2.6533798972 1.5333891366 -8.7316179193 1 1 1 7.9713478584 1.5258548152 -8.7275686163 1 1 1 -5.2901369599 6.1501470806 -8.7275606477 1 1 1 0.0020257219 6.1445633683 -8.6976678046 1 1 1 5.3019326567 6.1448522618 -8.7326135025 1 1 1 0.0000000000 0.0000000000 -13.0115119207 1 0 1 5.3119274980 0.0000000000 -13.0115119207 1 0 1 10.6238549960 0.0000000000 -13.0115119207 1 0 1 -2.6559637490 4.6002641563 -13.0115119207 1 0 1 2.6559637490 4.6002641563 -13.0115119207 1 0 1 7.9678912470 4.6002641563 -13.0115119207 1 0 1 -5.3119274980 9.2005283126 -13.0115119207 1 0 1 -0.0000000000 9.2005283126 -13.0115119207 1 0 1 5.3119274980 9.2005283126 -13.0115119207 1 0 1 -0.0000000000 3.0668427709 -17.3486825610 1 0 1 5.3119274980 3.0668427709 -17.3486825610 1 0 1 10.6238549960 3.0668427709 -17.3486825610 1 0 1 -2.6559637490 7.6671069272 -17.3486825610 1 0 1 2.6559637490 7.6671069272 -17.3486825610 1 0 1 7.9678912470 7.6671069272 -17.3486825610 1 0 1 -5.3119274980 12.2673710835 -17.3486825610 1 0 1 -0.0000000000 12.2673710835 -17.3486825610 1 0 1 5.3119274980 12.2673710835 -17.3486825610 1 0 1 &END &ESM BOUNDARY_CONDITION BARE &END
COOP計算の準備 †
- 入力ファイル (Total/nfinp_coop)
#
# CH3OH on Pt(111)
#
TASK COOP
WF_OPT DAV
NTYP 4
NATM 51
GMAX 6.0
GMAXP 20.0
KPOINT_MESH 4 4 1
WAY_MIX 6
MIX_ALPHA 0.5
SMEARING MP
WIDTH 0.02
EDELTA 1.D-12
NEG 374
&CELL
15.935782493970 0.000000000000 0.000000000000
-7.967891246985 13.800792468961 0.000000000000
0.000000000000 0.000000000000 65.057559603672
&END
&ATOMIC_SPECIES
Pt 195.084 pot.Pt_pbe1
H 1.00784 pot.H_lda1
C 12.0107 pot.C_pbe1
O 15.9994 pot.O_pbe1
&END
&ATOMIC_COORDINATES CARTESIAN
-1.8137028864 9.7577897649 6.0475265334 1 1 3
0.3796625950 9.4143384000 4.4413806754 1 1 4
-2.8866486133 7.9909878630 5.9662288609 1 1 2
1.3575317442 10.9863245180 4.4084228902 1 1 2
-3.0023990778 11.3232130902 5.3771267684 1 1 2
-1.1679860539 10.1062442042 7.9898686733 1 1 2
-0.0099652505 -0.0118152838 -0.0389267708 1 1 1
5.2888683372 0.0108921856 -0.0645303625 1 1 1
10.6265696551 0.0008317009 -0.0387744777 1 1 1
-2.6897091584 4.5448841905 -0.0523847489 1 1 1
2.6635463467 4.5580537376 -0.0706515335 1 1 1
7.9589507735 4.5854784695 -0.0722719517 1 1 1
-5.3637785701 9.1883685551 -0.0662604821 1 1 1
-0.0392907170 9.1565874594 0.0579966667 1 1 1
5.3281043692 9.1911533151 -0.0722007294 1 1 1
-0.0021214349 3.0614887173 -4.4301258499 1 1 1
5.3127589060 3.0682635440 -4.4675412941 1 1 1
10.6216354438 3.0680025298 -4.4594236536 1 1 1
-2.6410480936 7.6736054392 -4.3637257870 1 1 1
2.6463501164 7.6713174600 -4.3821208007 1 1 1
7.9673552854 7.6640546571 -4.4695568010 1 1 1
-5.3175076764 12.2702284809 -4.4320528195 1 1 1
0.0018447605 12.2611966334 -4.3749773596 1 1 1
5.3128659353 12.2716266162 -4.4267739348 1 1 1
0.0011760521 -3.0598724283 -8.7324837135 1 1 1
5.3220998470 -3.0659574817 -8.6963658587 1 1 1
10.6177996176 -3.0665582052 -8.6965151025 1 1 1
-2.6520476401 1.5342773535 -8.7269069007 1 1 1
2.6533798972 1.5333891366 -8.7316179193 1 1 1
7.9713478584 1.5258548152 -8.7275686163 1 1 1
-5.2901369599 6.1501470806 -8.7275606477 1 1 1
0.0020257219 6.1445633683 -8.6976678046 1 1 1
5.3019326567 6.1448522618 -8.7326135025 1 1 1
0.0000000000 0.0000000000 -13.0115119207 1 0 1
5.3119274980 0.0000000000 -13.0115119207 1 0 1
10.6238549960 0.0000000000 -13.0115119207 1 0 1
-2.6559637490 4.6002641563 -13.0115119207 1 0 1
2.6559637490 4.6002641563 -13.0115119207 1 0 1
7.9678912470 4.6002641563 -13.0115119207 1 0 1
-5.3119274980 9.2005283126 -13.0115119207 1 0 1
-0.0000000000 9.2005283126 -13.0115119207 1 0 1
5.3119274980 9.2005283126 -13.0115119207 1 0 1
-0.0000000000 3.0668427709 -17.3486825610 1 0 1
5.3119274980 3.0668427709 -17.3486825610 1 0 1
10.6238549960 3.0668427709 -17.3486825610 1 0 1
-2.6559637490 7.6671069272 -17.3486825610 1 0 1
2.6559637490 7.6671069272 -17.3486825610 1 0 1
7.9678912470 7.6671069272 -17.3486825610 1 0 1
-5.3119274980 12.2673710835 -17.3486825610 1 0 1
-0.0000000000 12.2673710835 -17.3486825610 1 0 1
5.3119274980 12.2673710835 -17.3486825610 1 0 1
&END
&ESM
BOUNDARY_CONDITION BARE
&END
&COOP
KPDOSMO_MOL_1 16
KATM_MOL_1 6
KLMTA_MOL_1 16
KPDOSMO_SUB 358
KATM_SUB 45
KLMTA_SUB 900
WFN_MOL_1 ../CH3OH/zaj.data
WFN_SUB ../Pt111/zaj.data
&END
以上の入力ファイルを用いてCOOP解析に必要な分子軌道および基板波動関数の全波動関数との重なり積分の計算を行います。この入力ファイルはSCF計算のものとほとんど同じですが、以下でCOOP計算の準備を指定します。
TASK COOP
また&COOP...&ENDブロックに
- KPDOSMO_MOL1(あるいはKPDOSMO1、吸着分子の軌道の数、非占有軌道も含めてある程度多めに)
- KATM_MOL_1(あるいはKATM1、吸着分子に含まれる原子数KATM_MOL_1)
- KLMTA_MOL_1(あるいはKLMTA1、分子の計算に必要な軌道角運動量と参照エネルギーの数)
- KPDOSMO_SUB(あるいはKPDOSMO3、基板のバンド数、非占有軌道も含めてある程度多めに)
- KATM_SUB(あるいはKATM3、基板に含まれる原子の数)
- KLMTA_SUB(あるいはKLMTA3、基板の計算に必要な軌道角運動量と参照エネルギーの数) を以下のように指定します。また
- WFN_MOL_1
- WFN_SUB
でそれぞれ分子および基板の波動関数のファイル名を指定します。
(デフォルトはそれぞれ./zak1.dataと./zak3.data)
&COOP KPDOSMO_MOL_1 16 KATM_MOL_1 6 KLMTA_MOL_1 16 KPDOSMO_SUB 358 KATM_SUB 45 KLMTA_SUB 900 WFN_MOL_1 ../CH3OH/zaj.data WFN_SUB ../Pt111/zaj.data &END
ここでKPDOSMO_MOL_1とKPDOSMO_SUBの和は全系のバンド数と一致させておく必要があります。 この入力ファイルを実行すると - coop.data
- coop_bij.data
- coop_sij.data が出力されます。またCOOPの計算の際にKohn-Sham固有値のファイル(eko.data)およびCOOP解析プログラム``coop_analysis``の入力ファイル``nfcoop.data``が出力されます(nfcoop.dataが存在する場合は上書きしない)。 ``nfcoop.data``は以下のような固定フォーマットになっています。
1 : KSPIN
10 : KNV3
8 16 6 16 : NPDOSMO1 KPDOSMO1 KATM1 KLMTA_1
0 0 0 0 : NPDOSMO2 KPDOSMO2 KATM2 KLMTA_2
358 358 45 900 : NPDOSMO3 KPDOSMO3 KATM3 KLMTA_3
-15.00 5.00 0.10 2001 : EMIN EMAX EWIDTH NPDOSE
COOP解析 †
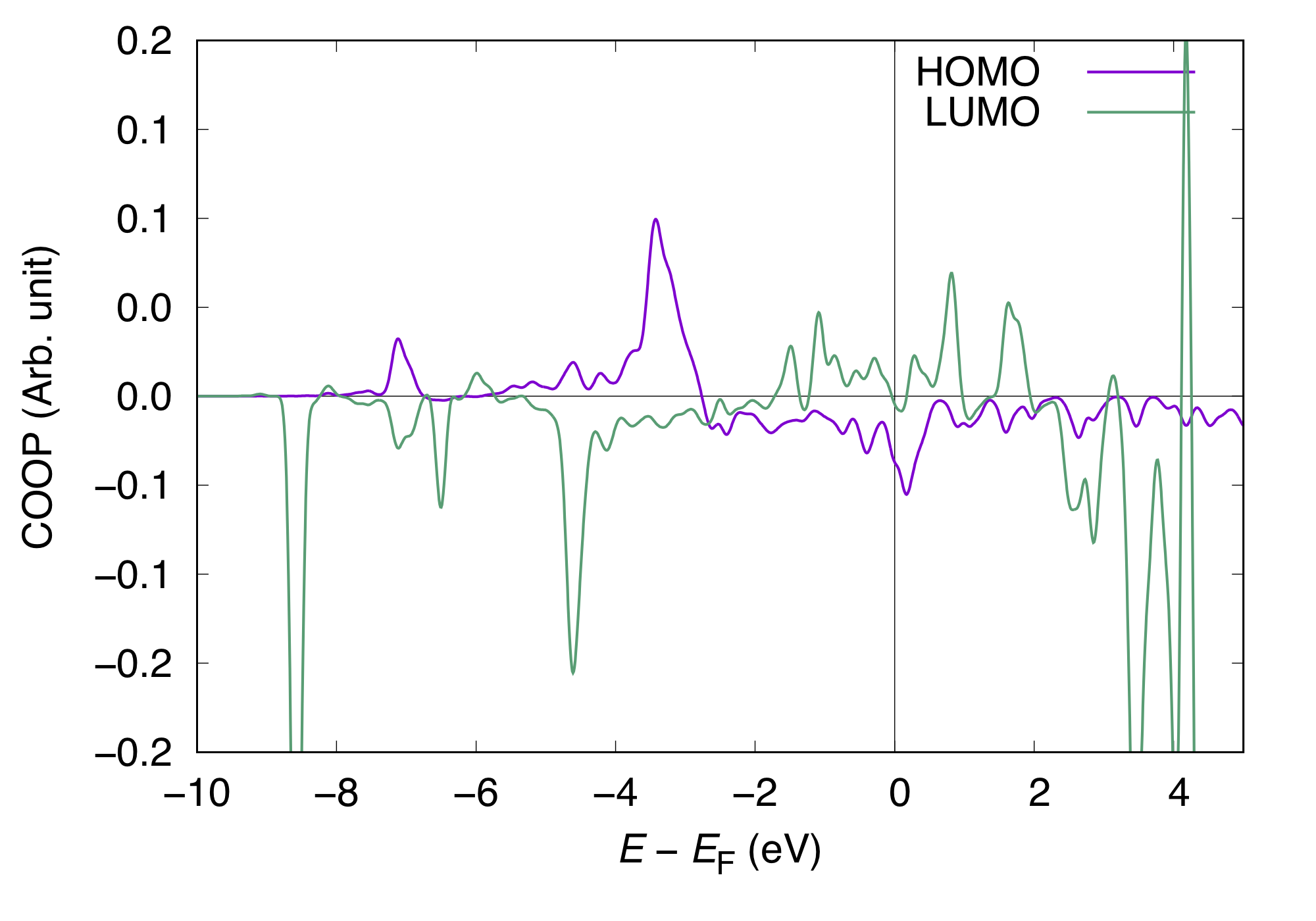
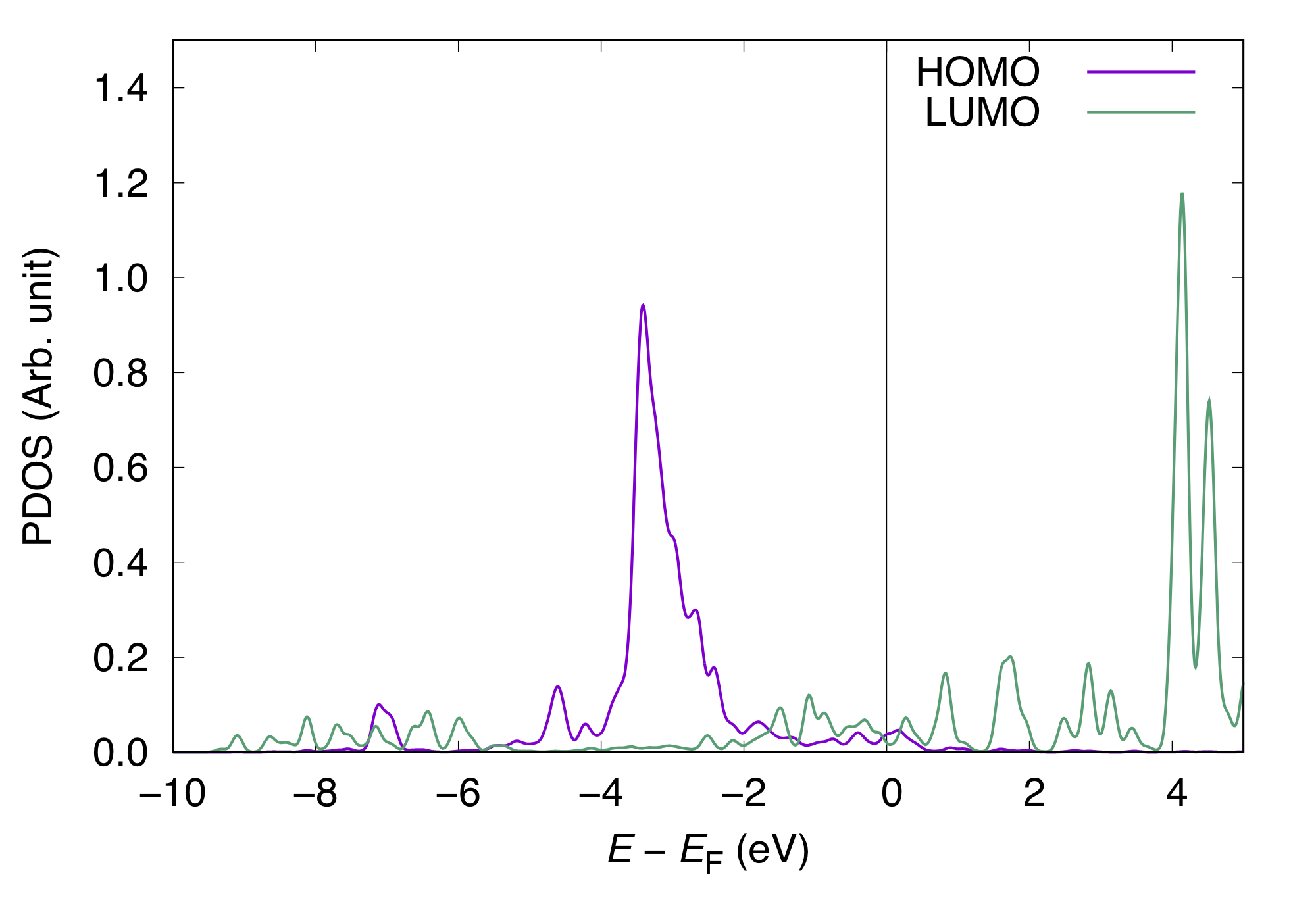
最後の``coop_analysis``を使ってCOOP解析を行います。 今の例で分子軌道として占有軌道 (7) および非占有軌道 (9) を考慮してオーバラップの計算を行いましたが、COOP解析の際に全部の分子軌道を利用することはありません。以下ではHOMOまで、およびLUMOまで考慮してCOOP解析を行ってみましょう。 (to be continued)
- KPDOSMO_MOL_1 (KPDOSMO1) = 8 この場合分子の軌道としてLUMOまでを考慮します。
- 入力ファイル (Total/nfcoop.data)
1 : KSPIN 10 : KNV3 8 16 6 16 : NPDOSMO1 KPDOSMO1 KATM1 KLMTA_1 0 0 0 0 : NPDOSMO2 KPDOSMO2 KATM2 KLMTA_2 358 358 45 900 : NPDOSMO3 KPDOSMO3 KATM3 KLMTA_3 -15.00 5.00 0.10 2001 : EMIN EMAX EWIDTH NPDOSE``coop_analysis``へのコマンドサーチパスが設定されていることを確認して以下を実行します。$ coop_analysis > coop_LUMO.out
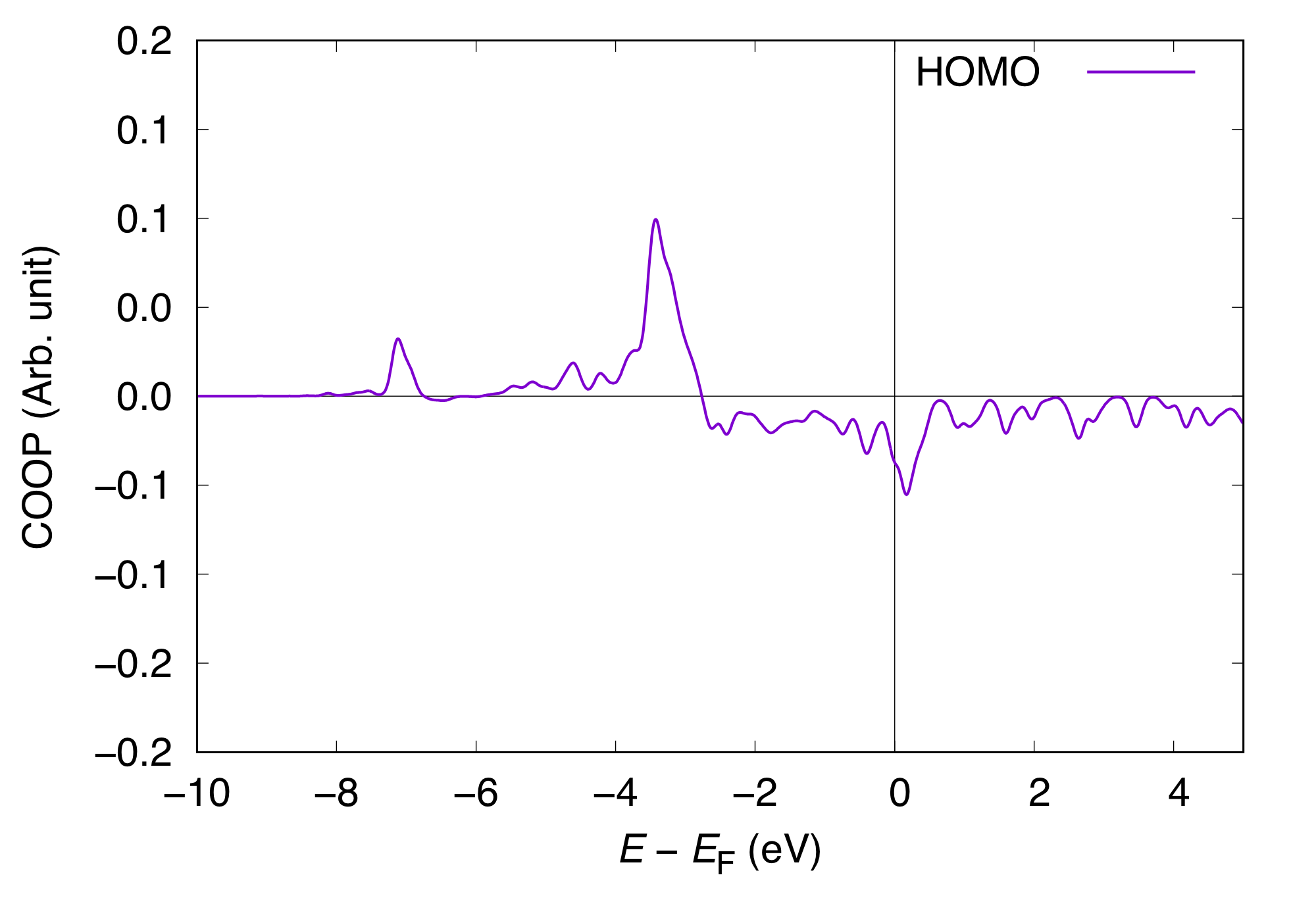
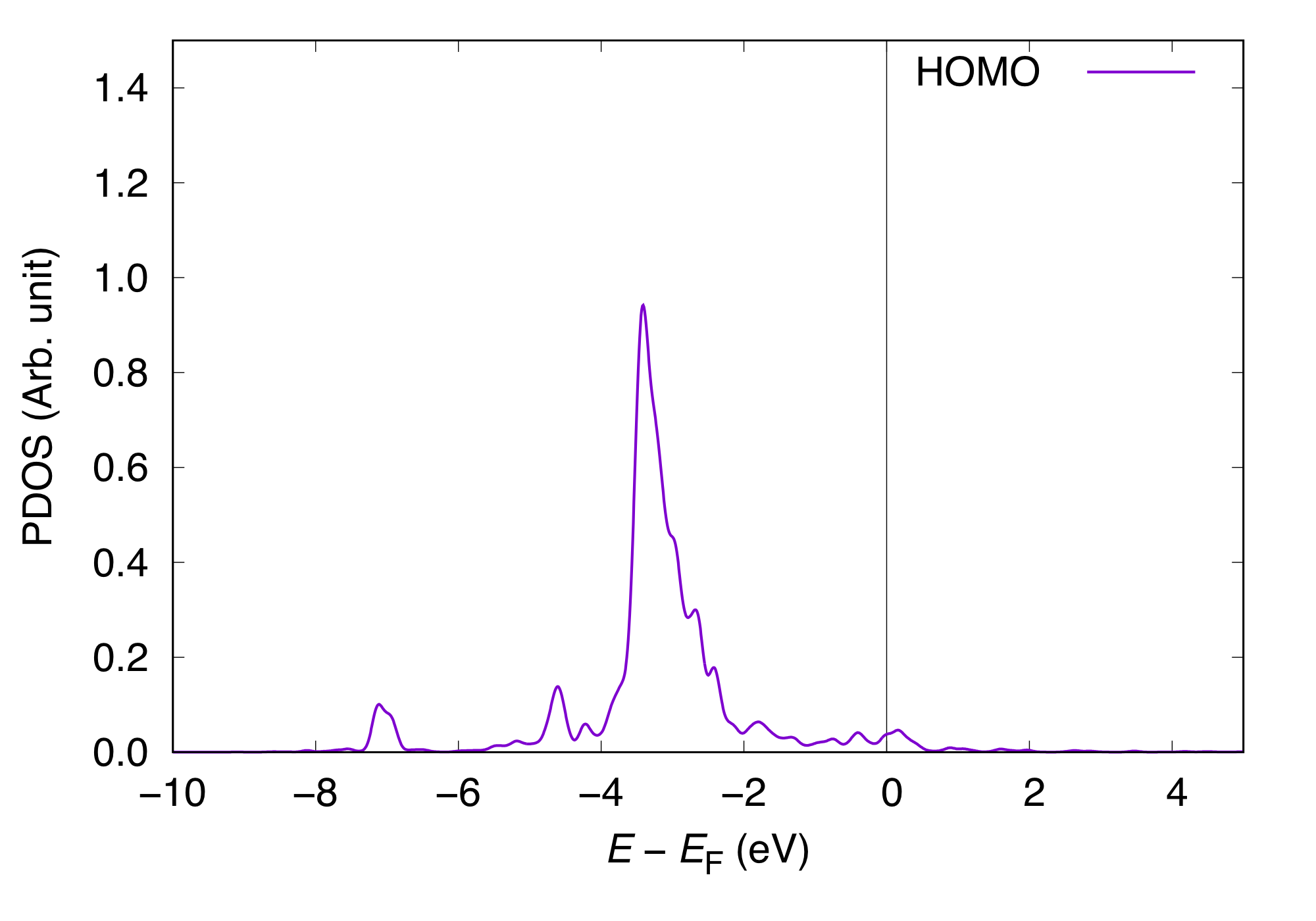
次に出力ファイルからCOOP (COOP2)のデータをgrepを使って取り出します。$ grep 'COOP2' coop_LUMO.out | awk -F\: '{print $2}' > coop2_LUMO.dat同様にして分子軌道に射影した状態密度 (MOPDOS) を取り出します。$ grep 'PDOS' coop_LUMO.out | awk -F\: '{print $2}' > pdos_LUMO.datこれらをgnuplotを使ってプロットします。HOMOとLUMOについてのCOOPとPDOSは以下のようになります。 COOP(正しくはCOOPの重みを付けた状態密度)は正の値が基板との結合的、負の値が反結合的相互作用を表しており、低いエネルギー領域ではCOOPは正になるべきですが、LUMOのCOOPは負の値から始まっています。PDOSもあわせてみるとLUMOは非占有状態(> Fermi energy)よりも大きいエネルギー領域に大きなピークを持ち、占有状態には大きなピークが存在しないことから占有状態にあるLUMOに由来するCOOPは非物理的であると考えられます。そのような場合は解析からLUMOの寄与を含めないのが良いでしょう。 - KPDOSMO_MOL_1 (KPDOSMO1) = 7 そのような考察のもと分子軌道としてはHOMOまでを考慮してもう一度coop_analysisを実行します。
- 入力ファイル (Total/nfcoop.data)
1 : KSPIN 10 : KNV3 7 16 6 16 : NPDOSMO1 KPDOSMO1 KATM1 KLMTA_1 0 0 0 0 : NPDOSMO2 KPDOSMO2 KATM2 KLMTA_2 358 358 45 900 : NPDOSMO3 KPDOSMO3 KATM3 KLMTA_3 -15.00 5.00 0.10 2001 : EMIN EMAX EWIDTH NPDOSE出力ファイル名前を以下のように変更して``coop_analysis``を実行します。$ coop_analysis > coop_HOMO.out
次に出力ファイルからCOOP (COOP2)のデータをgrepを使って取り出します。$ grep 'COOP2' coop_HOMO.out | awk -F\: '{print $2}' > coop2_HOMO.dat同様にしてMOPDOSを取り出します。$ grep 'PDOS' coop_HOMO.out | awk -F\: '{print $2}' > pdos_HOMO.datこの場合のHOMOとLUMOについてのCOOPとPDOSは以下のようになります。 LUMOを含めた場合とCOOP、PDOS共に形状はほぼ変わらないことが分かります。 またここでHOMOのCOOPの反結合性成分が部分的に占有されており、HOMOは結合的相互作用だけでなく基板と反結合的に相互作用していることも分かります。さらに差電荷の解析を行うことでそのような描像がより明かになります。
![[PukiWiki] [PukiWiki]](image/logo_state-senri3_80x80.png)