- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- STATE_new_tutorial/H2_OPT_jp へ行く。
- 1 (2021-07-29 (木) 11:08:34)
- 2 (2023-01-23 (月) 11:56:57)
- 3 (2023-08-22 (火) 19:01:42)
- 4 (2024-07-29 (月) 10:49:28)
水素分子 (構造最適化) †
ここでは分子の構造最適化の例として水素分子の平衡位置と平衡結合(ボンド)長を求めてみましょう。 現在のワーキングディレクトリが~/STATE/examplesであるとして、H2ディレクトリに移動します。
$ cd H2
ここでlsを実行し、nfで始まる入力ファイル群(nf*)や.shで終わるジョブスクリプト(*.sh)が含まれていることを確認します。 以下を実行して入力ファイルの例であるnfinp_scfを再確認しましょう。
$ cat nfinp_scf
- nfinp_scf
# H2 molecule
#
WF_OPT DAV
NTYP 1
NATM 2
GMAX 6.00
GMAXP 20.00
MIX_ALPHA 0.7
WIDTH 0.0010
EDELTA 0.1000D-09
NEG 2
CELL 10.00000000 10.00000000 10.00000000 90.00000000 90.00000000 90.00000000
&ATOMIC_SPECIES
H 2.000000 pot.H_pbe1_sp_new
&END
&INITIAL_ZETA
0.0000
&END
&ATOMIC_COORDINATES CARTESIAN
-0.699200000000 0.000000000000 0.000000000000 1 1 1
0.699200000000 0.000000000000 0.000000000000 1 1 1
&END
構造を確認するには以下のようにchkinpfを実行します。
$ chkinpf nfinp_scf
H2.xsfが出力されていることを確認して
$ xcrysden --xsf H2.xsf
を実行すると水素分子が大きな箱の中に配置されていることが分かります。
マニュアル構造最適化 †
先ずシリコンのセル最適化で行ったように水素分子の全エネルギーを最小化する配置を考えましょう。 気相の(孤立した)分子の計算を実行する際、単位胞は分子の大きさに対して十分大きく取り、分子の間の相互作用が無視できるようにする必要があります。水素分子については一辺の長さが10 Bohrの箱で十分であることが分かっていますので、原子位置のみ考慮します。 以下のように原子間距離(結合/ボンド長)を1.40 Bohrから1.50 Bohrまで0.01 Bohr刻みで変えた入力ファイルを作ってみましょう。
- nfinp_scf_1.40
#
# H2 molecule
#
WF_OPT DAV
NTYP 1
NATM 2
GMAX 6.00
GMAXP 20.00
MIX_ALPHA 0.7
WIDTH 0.0010
EDELTA 0.1000D-09
NEG 2
CELL 10.00000000 10.00000000 10.00000000 90.00000000 90.00000000 90.00000000
&ATOMIC_SPECIES
H 2.000000 pot.H_pbe1_sp_new
&END
&INITIAL_ZETA
0.0000
&END
&ATOMIC_COORDINATES CARTESIAN
0.000000000000 0.000000000000 0.000000000000 1 1 1
1.40 0.000000000000 0.000000000000 1 1 1
&END
- nfinp_scf_1.41
#
# H2 molecule
#
WF_OPT DAV
NTYP 1
NATM 2
GMAX 6.00
GMAXP 20.00
MIX_ALPHA 0.7
WIDTH 0.0010
EDELTA 0.1000D-09
NEG 2
CELL 10.00000000 10.00000000 10.00000000 90.00000000 90.00000000 90.00000000
&ATOMIC_SPECIES
H 2.000000 pot.H_pbe1_sp_new
&END
&INITIAL_ZETA
0.0000
&END
&ATOMIC_COORDINATES CARTESIAN
0.000000000000 0.000000000000 0.000000000000 1 1 1
1.41 0.000000000000 0.000000000000 1 1 1
&END
- ...
入力ファイルを確認することができたら以下のqsub_sb100.shの入出力ファイル名を変えてジョブを投入しましょう。
- qsub_sb100.sh
#$ -S /bin/sh
#$ -cwd
#$ -q sb.q
#$ -pe x6 6
#$ -N H2
module load intel/2021.2.0
module load intelmpi/2021.2.0
# Disable OPENMP parallelism
export OMP_NUM_THREADS=1
unset I_MPI_TCP_NETMASK
# Set the execuable of the STATE code
ln -fs ${HOME}/STATE/src/state-5.6.9/src/STATE .
# Set the pseudopotential data
ln -fs ../gncpp/pot.H_pbe1_sp_new
# Set the input/output file
INPUT_FILE=nfinp_scf
OUTPUT_FILE=nfout_scf
# Run!
mpirun ./STATE < ${INPUT_FILE} > ${OUTPUT_FILE}
得られた全エネルギーを結合長の関数としてetot.datというファイルにまとめましょう。
- etot.dat
1.40 -1.16767777 1.41 -1.16776031 1.42 -1.16780600 1.43 -1.16781613 1.44 -1.16779196 1.45 -1.16773476 1.46 -1.16764566 1.47 -1.16752570 1.48 -1.16737583 1.49 -1.16719699 1.50 -1.16699015
このデータをgnuplotで可視化してみましょう。
$ gnuplot
> plot 'etot.dat'
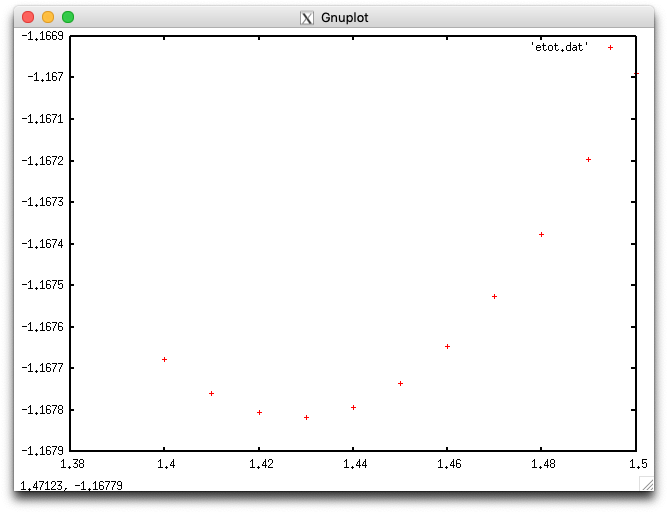
これを三次多項式でフィットすると以下のようになります。
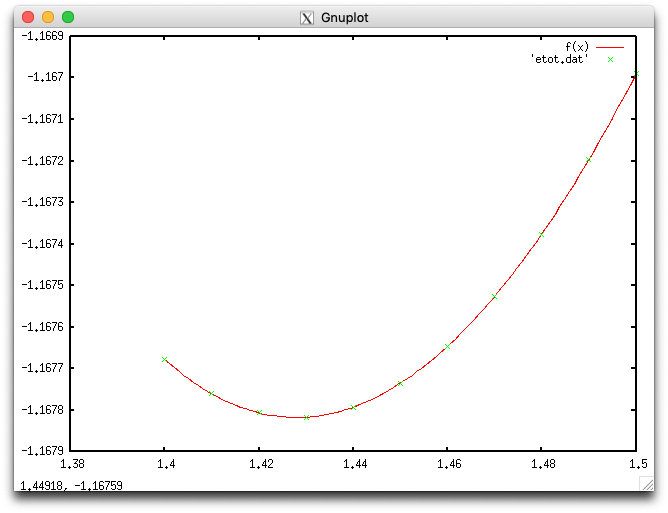
フィッティングから得られたパラメーターを用いて最小のエネルギーを与える結合長(平衡結合長)を求めると1.428 Bohrが得られます。
Hellmann-Feynman力を利用した構造最適化 †
マニュアル構造最適化は水素分子のような二原子分子では容易ですが、分子の自由度が増えてくると困難になります。一般的には原子にかかる力(Hellmann-Feynman力)を利用して構造最適化を行います。構造最適化には多くのアルゴリズムがありますが、ここではquenched molecular dynamics(QMD)を利用することにします。対応する入力ファイルをnfinp_qmdとします。
- nfinp_qmd
#
# H2 molecule
#
WF_OPT DAV
GEO_OPT QMD
FMAX 0.5000D-03
DTIO 10.00
NTYP 1
NATM 2
GMAX 6.00
GMAXP 20.00
MIX_ALPHA 0.7
WIDTH 0.0010
EDELTA 0.1000D-09
NEG 2
CELL 10.00000000 10.00000000 10.00000000 90.00000000 90.00000000 90.00000000
&ATOMIC_SPECIES
H 2.000000 pot.H_pbe1_sp_new
&END
&ATOMIC_COORDINATES CARTESIAN
-0.699200000000 0.000000000000 0.000000000000 1 1 1
0.699200000000 0.000000000000 0.000000000000 1 1 1
&END
ジョブスクリプト(qsub_sb100.sh)の入出力ファイル名を変更し(nfinp_qmd/nfout_qmd)、ジョブを投入します。 今の場合、系が単純なので計算が比較的早く終わります。 出力ファイル(nfout_qmd)をlessなどを使って見てみましょう。 SCF計算とは少し異なり、全エネルギーと原子にかかる力が出力された後に計算再開用データがrestart.dataに出力されたというメッセージが見られます。
ATOM COORDINATES FORCES MD: 1 MD: 1 H -0.699200 0.000000 0.000000 -0.01076 -0.00000 -0.00000 MD: 2 H 0.699200 0.000000 0.000000 0.01076 -0.00000 -0.00000 DQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQMDQ *I* restart data written in restart.data
restart.dataはバイナリーファイルですので人間の目で見ても分かりませんが、GEOMETRYというテキストファイルも構造最適化の各ステップで出力されますので、構造最適化がうまくいかなかった場合に新しい入力ファイルを作成する場合、GEOMETRYを利用すると良いでしょう。
この後に原子位置が更新され、再度SCF計算が行われます。 構造が自動で更新され、原子にかかる力(f_max)が入力ファイルで指定した閾値(FMAX 0.5000D-03 --> 5x10^-4 Bohr/Hartree)よりも小さくなったところで以下のメッセージを出力し計算が終了します。
CONVERGED ENERGY AND FORCES
NIT TotalEnergy f_max f_rms edel vdel fdel
15 -1.16781873 0.000484 0.000484 0.31D-11 0.14D-07 0.31D-11
ATOM COORDINATES FORCES
MD: 15
MD: 1 H -0.714657 -0.000000 -0.000000 0.00048 -0.00000 0.00000
MD: 2 H 0.714657 -0.000000 -0.000000 -0.00048 0.00000 0.00000
EXITING ATOM LOOP
最終的に得られた原子位置から結合長を計算すると1.429 Bohrが得られ、マニュアル構造最適化とフィッティングにより得られたものと良い一致を示していることが分かります。
![[PukiWiki] [PukiWiki]](image/logo_state-senri3_80x80.png)